本章主要介绍离散型朴素贝叶斯—— MultinomialNB 的实现。对于离散型朴素贝叶斯模型的实现,由于核心算法都是在进行“计数”工作、所以问题的关键就转换为了如何进行计数。幸运的是、Numpy 中的一个方法:bincount 就是专门用来计数的,它能够非常快速地数出一个数组中各个数字出现的频率;而且由于它是 Numpy 自带的方法,其速度比 Python 标准库 collections 中的计数器 Counter 还要快上非常多。不幸的是、该方法有如下两个缺点:
只能处理非负整数型中数组
向量中的最大值即为返回的数组的长度,换句话说,如果用 bincount 方法对一个长度为 1、元素为 1000 的数组计数的话,返回的结果就是 999 个 0 加 1 个 1
所以我们做数据预处理时就要充分考虑到这两点。
举个例子,当原始数据形如:
x, s, n, t, p, f
x, s, y, t, a, f
b, s, w, t, l, f
x, y, w, t, p, f
x, s, g, f, n, f
时,调用上述数值化数据的方法将会把数据数值化为:
0, 0, 0, 0, 0, 0
0, 0, 1, 0, 1, 0
1, 0, 2, 0, 2, 0
0, 1, 2, 0, 0, 0
0, 0, 3, 1, 3, 0
单就实现这个功能而言、实现是平凡的:
def quantize_data(x):
features = [set(feat) for feat in xt]
feat_dics = [{
_l: i for i, _l in enumerate(feats)
} if not wc[i] else None for i, feats in enumerate(features)]
x = np.array([[
feat_dics[i][_l] for i, _l in enumerate(sample)]
for sample in x])
return x, feat_dics
def get_prior_probability(self, lb=1):
return [(_c_num + lb) / (len(self._y) + lb * len(self._cat_counter))
for _c_num in self._cat_counter]
其中的 lb 为平滑系数(默认为 1、亦即拉普拉斯平滑),这对应的公式其实是带平滑项的、先验概率的极大似然估计:
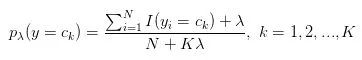
所以代码中的 self._cat_counter 的意义就很明确了——它存储着 K 个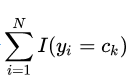
(cat counter 是 category counter 的简称)
同样先看核心实现:
data = [[]
for _ in range(n_dim)]for dim, n_possibilities in enumerate(self._n_possibilities):
data[dim] = [[
(self._con_counter[dim][c][p] + lb) / (
self._cat_counter[c] + lb * n_possibilities)
for p in range(n_possibilities)]
for c in range(n_category)]self._data = [np.array(dim_info) for dim_info in data]
这对应的公式其实就是带平滑项(lb)的条件概率的极大似然估计:
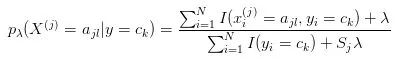
其中
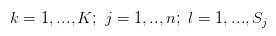
可以看到我们利用到了 self._cat_counter 属性来计算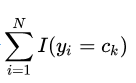 。同时可以看出:
。同时可以看出:
n_category 即为 K
self._n_possibilities 储存着 n 个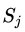
self._con_counter 储存的即是各个的值。具体而言: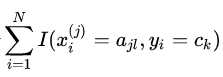
至于 self._data、就只是为了向量化算法而存在的一个变量而已,它将 data 中的每一个列表都转成了 Numpy 数组、以便在计算后验概率时利用 Numpy 数组的 Fancy Indexing 来加速算法
聪明的观众老爷可能已经发现、其实 self._con_counter 才是计算条件概率的关键,事实上这里也正是 bincount 大放异彩的地方。以下为计算 self._con_counter 的函数的实现:
def feed_sample_weight(self, sample_weight=None):
self._con_counter = []
for dim, _p in enumerate(self._n_possibilities):
if sample_weight is None:
self._con_counter.append([
np.bincount(xx[dim], minlength=_p) for xx in
self._labelled_x])
else:
self._con_counter.append([
np.bincount(xx[dim], weights=sample_weight[
label] / sample_weight[label].mean(), minlength=_p)
for label, xx in self._label_zip])
可以看到、bincount 方法甚至能帮我们处理样本权重的问题。
代码中有两个我们还没进行说明的属性:self._labelled_x 和 self._label_zip,不过从代码上下文不难推断出、它们储存的是应该是不同类别所对应的数据。具体而言:
self._labelled_x:记录按类别分开后的、输入数据的数组
self._label_zip:比 self._labelled_x 多记录了个各个类别的数据所对应的下标
这里就提前将它们的实现放出来以帮助理解吧:
# 获得各类别数据的下标
labels = [y == value for value in range(len(cat_counter))]
# 利用下标获取记录按类别分开后的输入数据的数组
labelled_x = [x[ci].T for ci in labels]
self._labelled_x, self._label_zip = labelled_x, list(zip(labels, labelled_x))
仍然先看核心实现:
def func(input_x, tar_category):
input_x = np.atleast_2d(input_x).T
rs = np.ones(input_x.shape[1])
for d, xx in enumerate(input_x):
rs *= self._data[d][tar_category][xx]
return rs * p_category[tar_category]
这对应的公式其实就是决策公式:
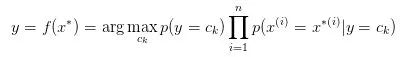
所以不难看出代码中的 p_category 存储着 K 个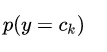
最后要做的、无非就是把上述三个步骤进行封装而已,首先是数据预处理:
def feed_data(self, x, y, sample_weight=None):
if sample_weight is not None:
sample_weight = np.array(sample_weight)
# 调用 quantize_data 函数获得诸多信息
x, y, _, features, feat_dics, label_dic = DataUtil.quantize_data(
x, y, wc=np.array([False] * len(x[0])))
# 利用 bincount 函数直接获得 self._cat_counter
cat_counter = np.bincount(y)
# 利用 features 变量获取各个维度的特征个数 Sj
n_possibilities = [len(feats) for feats in features]
# 获得各类别数据的下标
labels = [y == value for value in range(len(cat_counter))]
# 利用下标获取记录按类别分开后的输入数据的数组
labelled_x = [x[ci].T for ci in labels]
# 更新模型的各个属性
self._x, self._y = x, y
self._labelled_x, self._label_zip = labelled_x, list(
zip(labels, labelled_x))
self._cat_counter, self._feat_dics, self._n_possibilities = cat_counter, feat_dics, n_possibilities
self.label_dic = label_dic
self.feed_sample_weight(sample_weight)
然后利用上一章我们定义的框架的话、只需定义核心训练函数即可:
def _fit(self, lb):
n_dim = len(self._n_possibilities)
n_category = len(self._cat_counter)
p_category = self.get_prior_probability(lb)
data = [[] for _ in range(n_dim)]
for dim, n_possibilities in enumerate(self._n_possibilities):
data[dim] = [[
(self._con_counter[dim][c][p] + lb) / (
self._cat_counter[c] + lb * n_possibilities)
for p in range(n_possibilities)] for c in range(n_category)]
self._data = [np.array(dim_info) for dim_info in data]
def func(input_x, tar_category):
input_x = np.atleast_2d(input_x).T
rs = np.ones(input_x.shape[1])
for d, xx in enumerate(input_x):
rs *= self._data[d][tar_category][xx]
return rs * p_category[tar_category]
return func
最后,我们需要定义一个将测试数据转化为模型所需的、数值化数据的方法:
def _transfer_x(self, x):
for i, sample in enumerate(x):
for j, char in enumerate(sample):
x[i][j] = self._feat_dics[j][char]
return x
至此,离散型朴素贝叶斯就全部实现完毕了。
可以拿 UCI 上一个比较出名(简单)的“蘑菇数据集(Mushroom Data Set)”来评估一下我们的模型。该数据集的大致描述如下:它有 8124 个样本、22 个属性,类别取值有两个:“能吃”或“有毒”;该数据每个单一样本都占一行、属性之间使用逗号隔开。选择该数据集的原因是它无需进行额外的数据预处理、样本量和属性量都相对合适、二类分类问题也相对来说具有代表性。更重要的是,它所有维度的特征取值都是离散的、从而非常适合用来测试我们的MultinomialNB 模型。
完整的数据集可以参见这里(第一列数据是类别),我们的模型在其上的表现如下图所示:
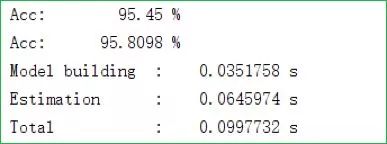
其中第一、二行分别是训练集、测试集上的准确率,接下来三行则分别是建立模型、评估模型和总花费时间的记录。
当然,仅仅看一个结果没有什么意思、也完全无法知道模型到底干了什么。为了获得更好的直观,我们可以进行一定的可视化,比如说将极大似然估计法得到的条件概率画出(如第零章所示的那样)。可视化的代码实现如下:
# 导入 matplotlib 库以进行可视化
import matplotlib.pyplot as plt
# 进行一些设置使得 matplotlib 能够显示中文
from pylab import mpl
# 将字体设为“仿宋”
mpl.rcParams['font.sans-serif'] = ['FangSong']
mpl.rcParams['axes.unicode_minus'] = False
# 利用 MultinomialNB 搭建过程中记录的变量获取条件概率
data =nb["data"]
# 定义颜色字典,将类别 e(能吃)设为天蓝色、类别 p(有毒)设为橙色
colors = {"e": "lightSkyBlue", "p": "orange"}
# 利用转换字典定义其“反字典”,后面可视化会用上
_rev_feat_dics = [{_val: _key for _key, _val in _feat_dic.items()}
for _feat_dic in self._feat_dics]
# 遍历各维度进行可视化
# 利用 MultinomialNB 搭建过程中记录的变量,获取画图所需的信息
for _j in range(nb["x"].shape[1]):
sj = nb["n_possibilities"][_j]
tmp_x = np.arange(1, sj+1)
# 利用 matplotlib 对 LaTeX 的支持来写标题,两个 $ 之间的即是 LaTeX 语句
title = "$j = {}; S_j = {}$".format(_j+1, sj)
plt.figure()
plt.title(title)
# 根据条件概率的大小画出柱状图
for _c in range(len(nb.label_dic)):
plt.bar(tmp_x-0.35*_c, data[_j][_c, :], width=0.35,
facecolor=colors[nb.label_dic[_c]], edgecolor="white",
label="class: {}".format(nb.label_dic[_c]))
# 利用上文定义的“反字典”将横坐标转换成特征的各个取值
plt.xticks([i for i in range(sj + 2)], [""] + [_rev_dic[i] for i in range(sj)] + [""])\
plt.ylim(0, 1.0)
plt.legend()
# 保存画好的图像
plt.savefig("d{}".format(j+1))
由于蘑菇数据一共有 22 维,所以上述代码会生成 22 张图,从这些图可以非常清晰地看出训练数据集各维度特征的分布。下选出几组有代表性的图片进行说明。
一般来说,一组数据特征中会有相对“重要”的特征和相对“无足轻重”的特征,通过以上实现的可视化可以比较轻松地辨析出在离散型朴素贝叶斯中这两者的区别。比如说,在离散型朴素贝叶斯里、相对重要的特征的表现会如下图所示(左图对应第 5 维、右图对应第 19 维):
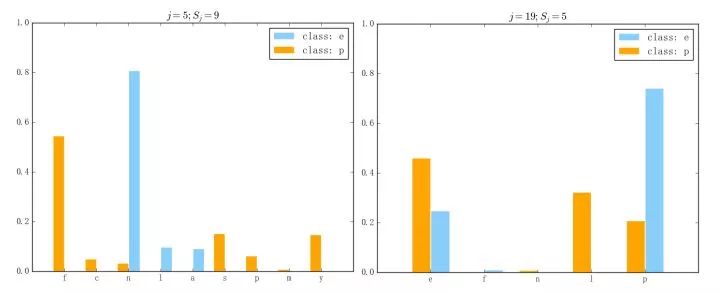
可以看出,蘑菇数据集在第 19 维上两个类别各自的“优势特征”都非常明显、第 5 维上两个类别各自特征的取值更是基本没有交集。可以想象,即使只根据第 5 维的取值来进行类别的判定、最后的准确率也一定会非常高
那么与之相反的、在 MultinomialNB 中相对没那么重要的特征的表现则会形如下图所示(左图对应第 3 维、右图对应第 16 维):
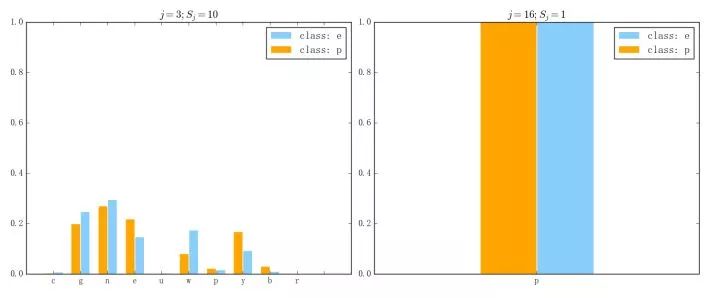
可以看出,蘑菇数据集在第 3 维上两个类的特征取值基本没有什么差异、第 16 维数据更是似乎完全没有存在的价值。像这样的数据就可以考虑直接剔除掉。